51直播 新闻
Genome Biol | 杨恩策团队揭示转座元件起始转录本的表达调控和进化规律
51直播
转座元件(Transposable elements, TEs)是广泛存在于真核生物基因组中的可移动遗传元件,约占人类基因组的45%[1]。由于具备转座能力且富含调控序列,TE被认为是宿主基因组调控元件进化的重要驱动力,能够在较短的时间尺度上引发显著变化[2]。尽管多数TE因突变和表观遗传机制而被沉默,但越来越多的研究表明,部分TE仍保持转录活性,并在胚胎发育、免疫反应及组织稳态中发挥作用[3]。其中,作为替代启动子,TE可驱动自身或邻近基因的转录,产生TE起始转录本[4]。然而由于TE的重复元件特性,其在短读长RNA-seq数据中易产生模糊比对,限制了对其表达规律和调控机制的系统解析。
2025年11月4日,51直播-51直播网 杨恩策团队在Genome Biology发表题为Transposable elements drive species-specific and tissue-specific transcriptomes in human development的论文。文章系统整合了多种类型的转录组学数据,开发了高精度识别TE起始转录本的生物信息学工具和流程,通过系统性识别人体生理组织部位和胚胎干细胞的TE起始转录本,揭示了TE作为演化元件池,在长期演化过程中通过转座事件为基因提供了广泛的可变转录起始位点,并通过其具有的多样的转录因子结合位点,组织特异性的塑造了灵长类的基因表达网络,为同源基因转录分化的精细调控提供了新见解。

在该工作中,研究者首先通过多种转录组学数据的整合实现了TE起始转录本的准确鉴定。通过对生理条件下TE起始转录本的系统鉴定,这些TE起始转录本不仅呈现出明显的组织特异性表达的特征,且其嵌合表达基因功能显著富集于对应组织的生物学过程。
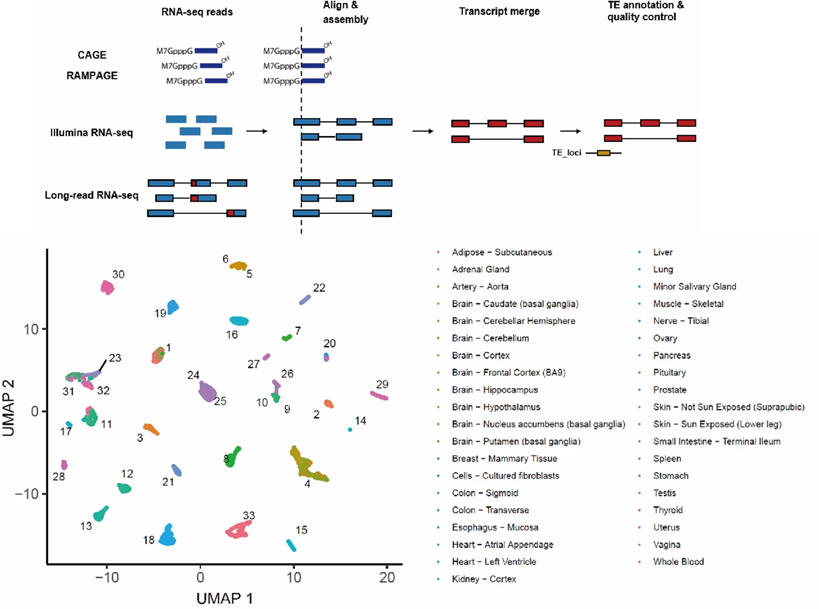
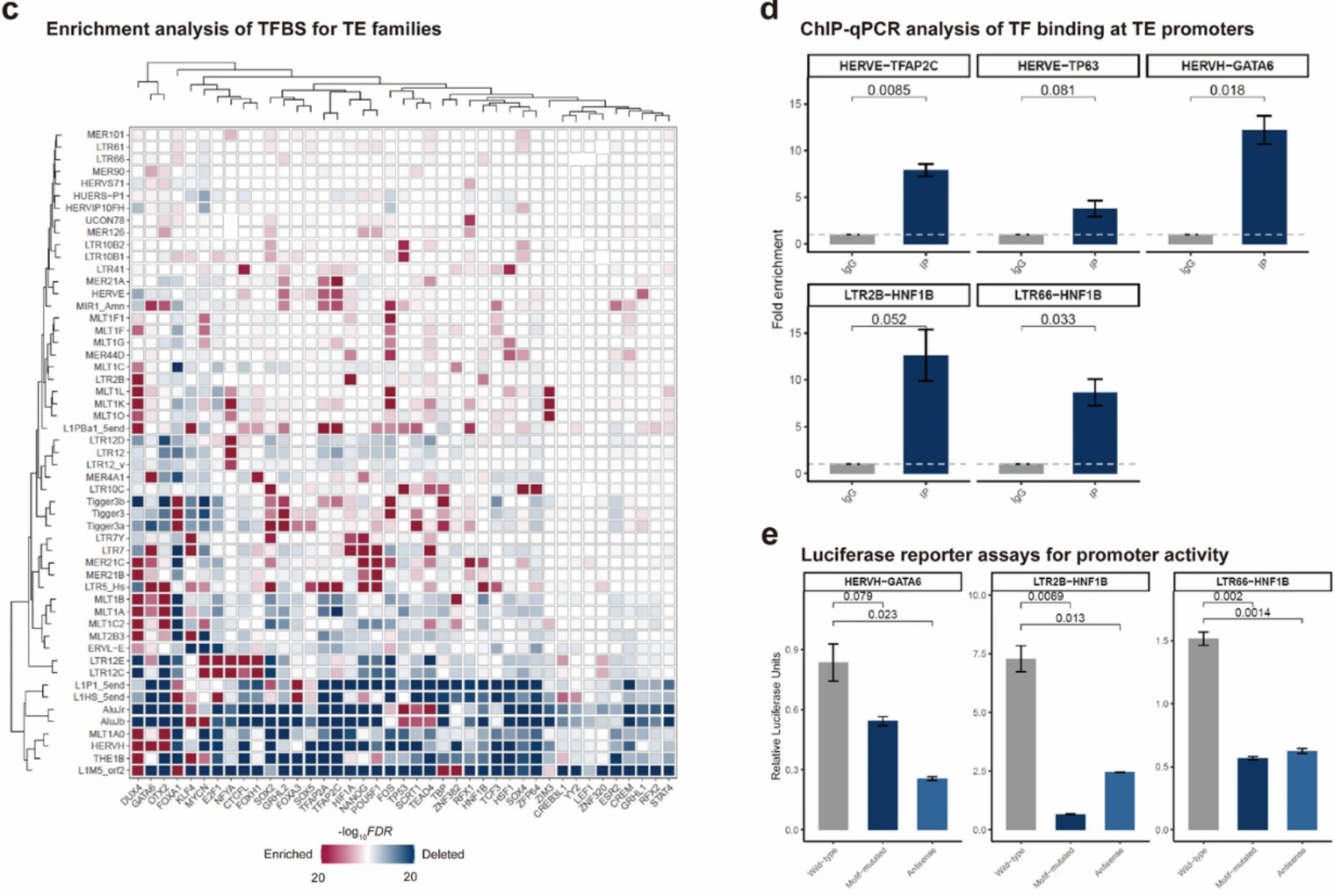
进一步对组织特异性来源的分析显示TE家族富集有表达对应的组织特异性转录因子结合基序。例如,LTR7和HERVE的转座分别产生了数百个新的包含NANOG和TFAP2C结合基序的TE启动子,参与胚胎干细胞干性维持和皮肤角质细胞成熟过程的基因表达调控。细胞系的ChIP-qPCR和荧光素酶报告实验验证了识别基序的结合能力。
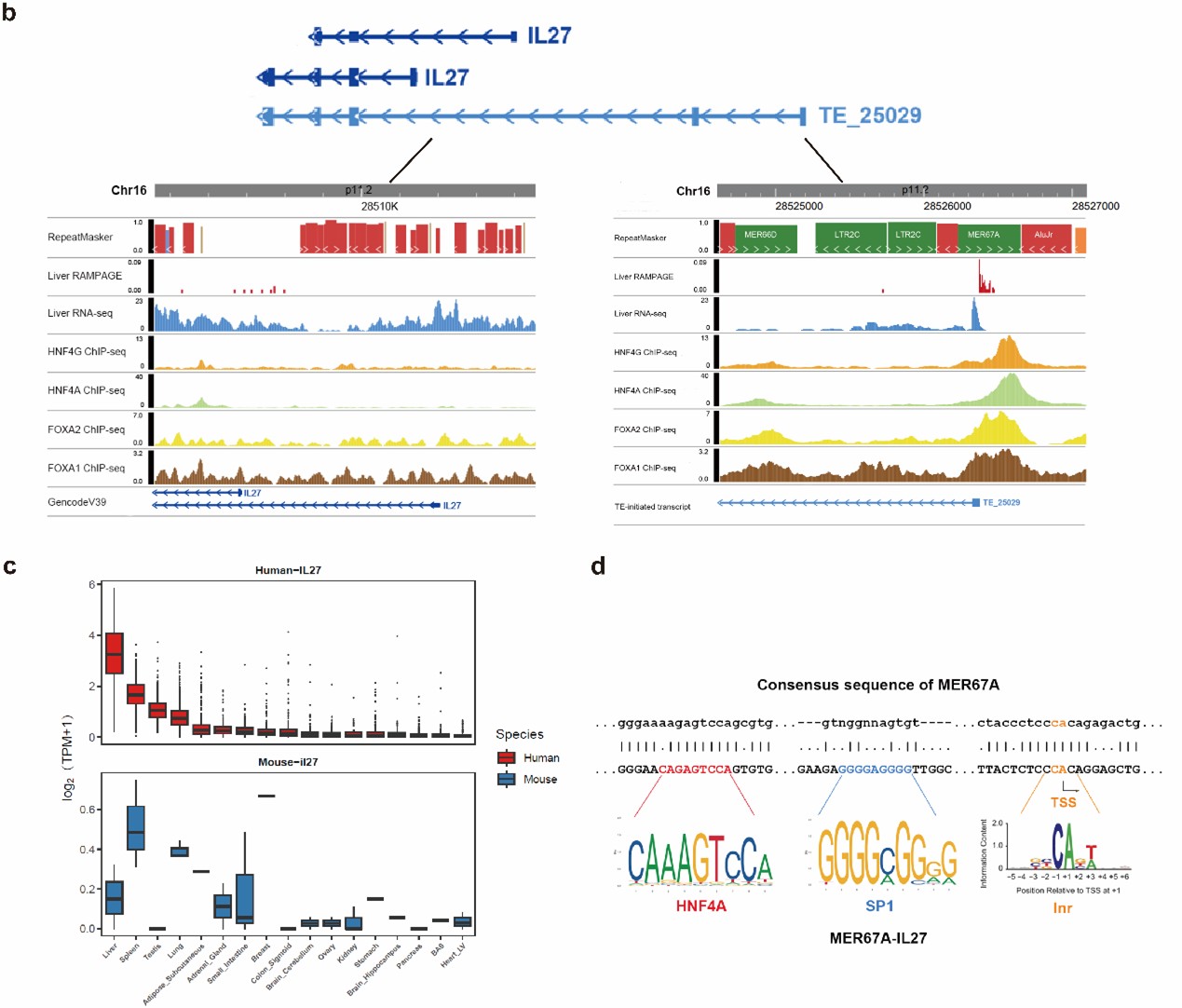
由于灵长类漫长的进化过程中具有多轮TE插入。因此,研究进一步分析了TE起始转录本的表达保守性,发现活跃转录确实与TE年龄相关,并鉴定出了312个包含灵长类特异TE转录起始位点,且组织特异性表达模式发生显著变化的人鼠同源基因。例如人类IL27基因上游MER67A元件的插入,后续的基因突变使其形成了一个新的TE启动子,该启动子包含HNF4A转录因子结合基序,可驱动IL27基因的肝组织特异性表达,而在小鼠肝脏中该基因几乎不表达。并且,该基因的表达变化可能与灵长类肝脏的免疫调节和能量代谢过程有关。
综上,该研究在多个人体组织中进行了TE起始转录本的系统鉴定,发现大多数转录本呈现组织特异性表达模式,并与TE介导的组织特异性转录因子结合位点扩散密切相关,其中约一半的TE起始转录本为灵长类特有,提示驯化的TE可以作为灵长类动物特异性的替代启动子,改变基因表达的器官特异性。本研究系统性回答了TE在灵长类组织特异性基因调控网络塑造中具有的重要作用,为理解人类基因组进化及组织特异性功能调控机制提供了新的证据。
51直播-51直播网 张赟博士和佘坚祺博士后为本文的共同第一作者,51直播-51直播网 杨恩策研究员为本文的通讯作者。
原文链接://doi.org/10.1186/s13059-025-03855-5
参考文献:
[1] Wells JN, Feschotte C. A field guide to eukaryotic transposable elements. Annu Rev Genet. 2020;21(54):539.
[2] Andrews G, Fan K, Pratt HE, Phalke N, Zoonomia Consortium§, Karlsson EK, et al. Mammalian evolution of human cis-regulatory elements and transcription factor binding sites. Science. 2023;380(6643):eabn7930.
[3] Pasquesi GIM, Perry BW, Vandewege MW, Ruggiero RP, Schield DR, Castoe TA. Vertebrate lineages exhibit diverse patterns of transposable element regulation and expression across tissues. Genome Biol Evol. 2020;12(5):506–21.
[4] Shah NM, Jang HJ, Liang Y, Maeng JH, Tzeng SC, Wu A, et al. Pan-cancer analysis identifies tumor-specific antigens derived from transposable elements. Nat Genet. 2023;55(4):631–9.

